南科大靳文菲课题组开发组学大数据素描压缩方法
快科技:近日,南方科技大学生命科学学院生物系副教授靳文菲课题组开发了组学大数据素描压缩方法实现组学大数据实时处理。该成果以“Kssd: Sequence Dimensionality-Reduction by K-mer Substring Space Sampling Enables Real-time Large-Scale Dataset Analysis”为题发表在基因组学领域知名学术杂志Genome Biology。
DNA测序技术的进步带来了测序通量的快速增长和测序成本的快速降低。伴随着DNA测序技术的广泛应用,各类人群基因组项目的实施,以及组学数据进一步激增,多个生物医学数据库里已有海量组学数据,如美国国家生物技术信息中心(National Center for Biotechnology Information, 简称NCBI)的SRA数据库的数据已超过30PB(3X1015)。虽然组学大数据为我们进行各种数据挖掘提供了丰富的资源,但是处理组学大数据对现有的存储和计算带来巨大挑战。多数实验室因为没有足够的存储和计算能力而无法利用组学大数据开展研究,严重阻碍了对数据生物学意义的解读。为促进对组学大数据的研究,研究小组开发了一种组学大数据素描方法(data sketching)对数据压缩降维,命名为子序列空间分解(k-mer substring space decomposition;Kssd)。Kssd可将序列大数据(包括基因组、转录组、宏基因组)降维几千至几十万倍之后仍能保持数据集之间距离或者相似度不变。因此实现了超大规模组学大数据的实时分析(如聚类、质控、搜索等)。该研究同时分析了NCBI SRA公共数据库的1023960个细菌基因组序列数据集以及1730个千人基因组计划数据集并发现了其中的误标样本。
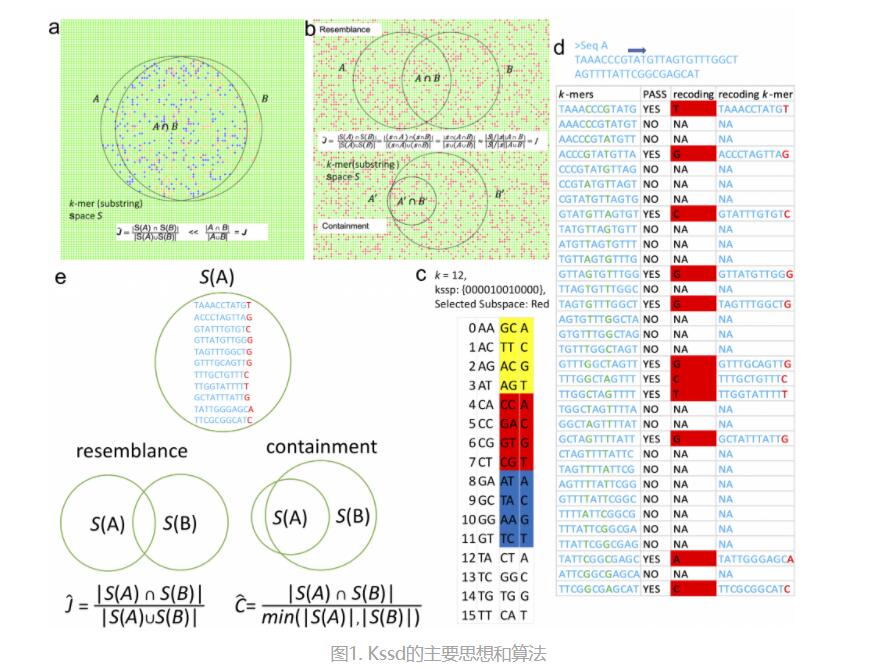
图1. Kssd的主要思想和算法
研究团队把每一条序列看成是固定长度的k-mer组成的集合(k-mer是将一条序列分成包含k个碱基的子字符串)。在每个k-mer集合中随机抽出一个k-mer子集,当满足两序列间的相似度等于其k-mer子集间的相似度,这个k-mer子集被称为该序列的素描(sketch)而序列降维被称为序列素描法(sketching)。目前主要的序列素描方法Minhash通过选取n(n取值一般在1000左右)个哈希值最小的k-mer做为k-mer子集。当两个序列大小相差悬殊时,Minhash法抽取的k-mer子集间的相似度将偏离两序列的相似度。为了做到无偏素描,该研究提出了K-mer全空间抽样的思想,即先在K-mer全空间上抽取K-mer子集,把待降维的序列的K-mer集与该抽取K-mer子集的交集作为该序列的素描。这种方法构建的序列素描,即使在两序列大小相差悬殊的情况下,也能准确度量两序列之间的相似度,从而拓展了序列降维方法的应用范围。
该研究方法进一步实现了序列素描之间的集合运算(包括交、并、减等运算),并发现人群体基因组序列的素描在减除参考基因组序列素描之后得到的余数基因组素描,可用以进行快速而精确的群体基因组大数据集聚类。而目前其他序列素描方法尚无法在群体水平上对序列进行精确聚类。该方法应用领域广泛,适用于任何需要对序列之间的相似性进行近似估计的应用,比如构建进化树、宏基因组成分分析及大规模数据集搜索等等,是对生物信息学领域基础方法学的一次重大创新。
靳文菲课题组博士后易会广为论文第一作者,南科大为第一署名单位,靳文菲为通讯作者。2020届南科大-哈工大硕士生毕业生林彦灵和东南大学教授林承棋参与了该工作。研究得到国家重点研发计划、国家自然科学基金、深圳市科技创新委员会、中国博士后科学基金以及江苏省博士后科研基金的资助。(来源:南方科技大学)